Scienziati della University of Illinois at Urbana-Champaign hanno elaborato una simulazione 3D che replica una cellula vivente, utilizzando processori grafici di Nvidia.
Ogni cellula vivente contiene un suo vivace microcosmo, con migliaia di componenti responsabili della produzione di energia, della costruzione di proteine, della trascrizione dei geni e altro ancora.
Gli scienziati americani, nella simulazione 3D, hanno replicato queste caratteristiche fisiche e chimiche su scala di particelle, creando un modello completamente dinamico che imita il comportamento di una cellula vivente.
Pubblicato sulla rivista Cell, il progetto simula una cellula minimale vivente, che contiene una serie ridotta di geni essenziali per la sopravvivenza, la funzione e la replicazione della cellula.
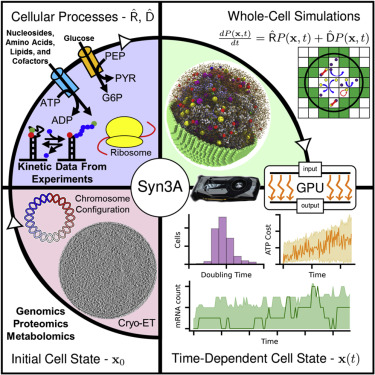
Il modello utilizza GPU Nvidia per simulare 7.000 processi di informazione genetica su un arco di 20 minuti del ciclo cellulare.
Ciò la ha resa quella che gli scienziati ritengono essere la più lunga e complessa simulazione cellulare fino ad oggi.
Le cellule minimali sono più semplici di quelle naturali, il che le rende più facili da ricreare digitalmente.
Tuttavia – sottolineano gli scienziati – anche una cellula minimale richiede due miliardi di atomi, e non è possibile fare un modello 3D come questo in una scala temporale umana realistica senza le GPU.
Una volta ulteriormente testati e raffinati, i modelli di cellule intere – ha messo in evidenza Nvidia – possono aiutare gli scienziati a prevedere come le modifiche alle condizioni o ai genomi delle cellule del mondo reale influenzeranno la loro funzione.
Ma anche in questa fase, la simulazione cellulare minimale può dare agli scienziati una visione dei processi fisici e chimici che formano la base delle cellule viventi.

Lattice Microbes, il software accelerato da GPU co-sviluppato da Luthey-Schulten e utilizzato per simulare la cellula minimale 3D, è disponibile sull'hub software Nvidia NGC.
Utilizzando il software Lattice Microbes su GPU Nvidia Tensor Core, i ricercatori hanno eseguito una simulazione 3D di 20 minuti del ciclo di vita della cellula, prima che inizi a espandersi sostanzialmente o a replicare il suo DNA.
L’autore principale dell'articolo Zane Thornburg sta lavorando a un altro progetto accelerato dalle GPU, per simulare la crescita e la divisione cellulare in 3D.
Il team ha recentemente adottato i sistemi Nvidia DGX e le GPU RTX A5000 per accelerare ulteriormente il lavoro.
E ha scoperto che l'uso delle GPU A5000 ha accelerato il tempo di simulazione del benchmark del 40% rispetto a una workstation di sviluppo con una GPU Nvidia di generazione precedente.